You’ve asked for it, darling. Ick geh den Query mal Schritt für Schritt durch (wer nicht Flux lernen will, sollte hier vmtl. mit dem Lesen abbrechen.)
jan = from(bucket: "dwd_cdc")
Wir nehmen uns mal die “dwd_cdc”-Datenbank, aaaber tun das Ergebniss dieses ja noch weiter spezifizierten Queries in eine Art Variable und nennen sie “jan”.
|> range(start:2019-01-01T00:00:00Z, stop: 2019-01-31T23:59:59Z)
Joar, hier würde mensch sonst[tm] “$range” in die Klammern schreiben: Eine Variable, die Grafana anhand des aktuell gewählten Zeitfensters Flux-gerecht bereitstellt. Aber wir wollen ja mal nur den Januar betrachten. Unsere DB denkt in UTC, der DWD auch: Schick.
|> filter(fn: (r) =>
r._measurement == "dwd_cdc_temp_2m_c" and
r._field == "value" and
r.sta_name == "$COMMON_CDC_NAME" and
r.quality_level == "2" and
r.produkt == "10_minutes"
)
Lasst uns unsere Datenbank filtern! “r” definieren wir als unsere response. “interne” Variablen beginnen mit nem Undersore; das ist für _measurement, _field und _value gegeben. Übrige/individuelle tags werden ohne
_ angegeben. Der Wert aus der Variable $CDC_COMMON_NAME stellt das Grafana über unser Station-Dropdown bereit (hier könnte auch nen Stationsname “hardcodet” stehen).
|> aggregateWindow(every: 1d, fn: mean)
Ist ne Helper-Function, die eigentlich erstmal window() aufruft, und denn die einzelnen Series wieder group()t. Damit lassen sich auch verschiedene Messrhytmen “normalisieren” und vergleichen. In diesem Fall machen wir aus unseren “alle 10min-Daten” die gewünschten Tagesmittelwerte.
|> keep(columns: ["_value", "_time"])
Lasst uns unnötiges Wegschmeißen.
|> filter(fn: (r) => r._value > 0)
Ah, wir interessieren uns ja nur für Tagesmittelwerte über 0°C! Also filtern wir mal auf die. Das macht ja auch jetzt erst Sinn, weil wir vorher noch nicht auf Tagesmittelwerte aggregiert haben.
|> map(fn: (r) => ({
_time: r._time,
_value: r._value * 0.5
}))
Tja, dann müssen wa noch unseren Wert gewichten. Ist ja Januar, also mit 0.5. _time muss mit aufgerufen werden, damits überlebt.
Ok, dasselbe für
feb = from(bucket: "dwd_cdc")
und analog für
rest = from(bucket: "dwd_cdc")
Jetz dieses union():
union(tables: [jan, feb, rest])
|> sort(columns: ["_time"], desc: false)
Sortierung ist wichtig, sonst kommt etwa der 1.-11. Februar zuerst in die Tabelle und dananch erst der 1.-31. Januar und das arme Grafana ist sehr verwirrt und malt plötzlich Kreise. (Srsly. Grafana supports time-travel!)
|> map(fn: (r) => ({
_time: r._time,
_value: r._value,
foo: "GTS Tag"
}))
Boar, diese Beschriften von einzelnen Datenserien ist die Hölle mit Grafana & Flux. Hier fügen wir an jeden Messwert noch nen key-value-Päarchen, damit wir die Serie als “Tag”(eswert) wiedererkennen können.
|> yield(name: "bars")
Weil wir hier grafisch Balken nehmen, heissts “bars” und es wird die Series an genau dieser Stelle per yield() “vermeldet”, also als separate result-Tabelle ausgeworfen und gemalt, ehe wir sie weiter modizifieren. Somit haben wir jetz erstmal alle gewichteten Tagesmittelwerte > 0°C im Bild.
|> cumulativeSum(columns: ["_value"])
Wir wollen aber auch die kummulierte Summe aller Werte! (Bei single stats: Nur sum() )
|> map(fn: (r) => ({
_time: r._time,
_value: r._value,
foo: "GTS Total"
}))
Also benennen wir unser lustiges Feld nochmal um und brauchen es nicht nochmal yield(en), weil wir jetzt ja am Ende sind und damit eh unsere/(die) eine Ergebnistabelle rendern.
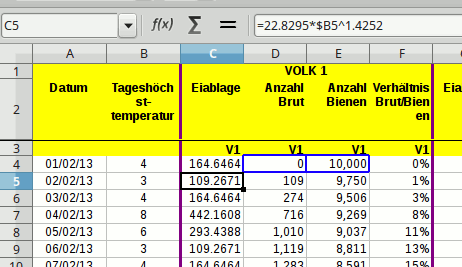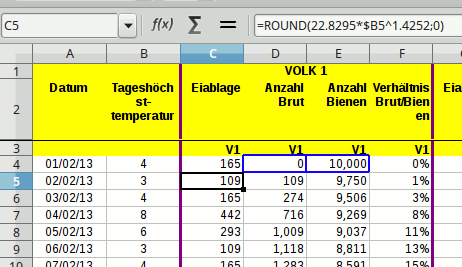
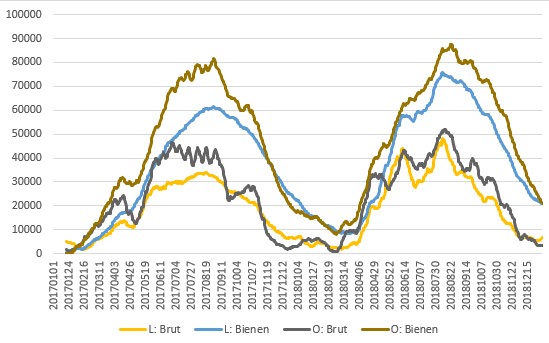
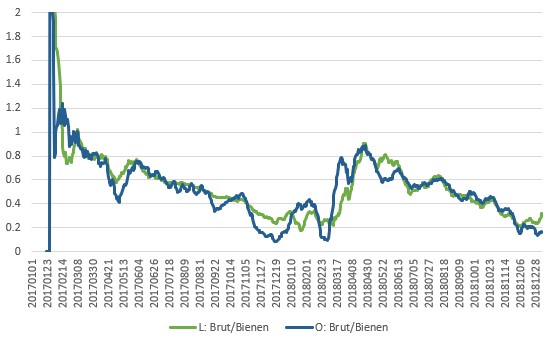
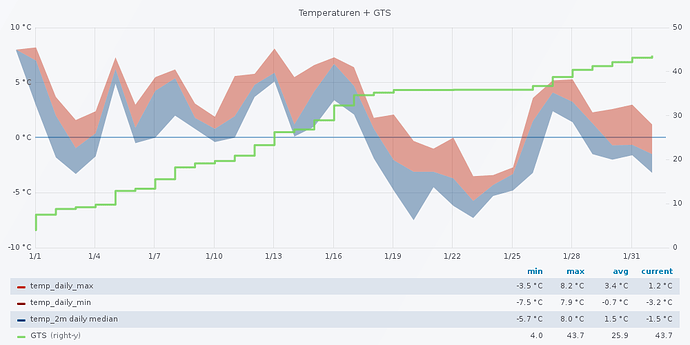
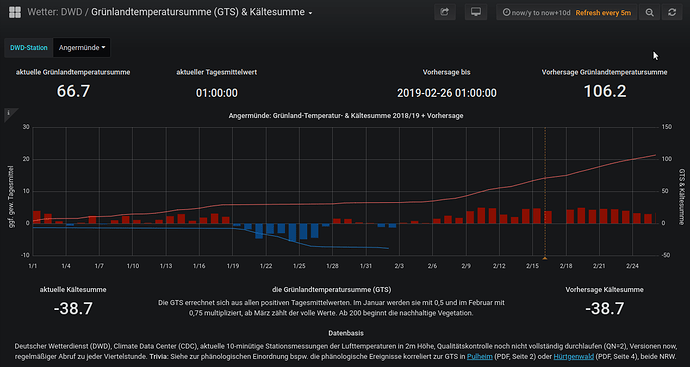
 … ja, das sind dann auch nur im positiven je Zeitraum drei und für die Vorhersage je nochmal 3x2 = 6 Teilabfragen in einen Query (Kältesumme läuft separat). [Edit: Ein Glück müssen wa nun nicht mehr für die zwei Darstellungen (je Tag vs. akkumulierten Gesamtwert) noch einen Satz Queries fahren. :]
… ja, das sind dann auch nur im positiven je Zeitraum drei und für die Vorhersage je nochmal 3x2 = 6 Teilabfragen in einen Query (Kältesumme läuft separat). [Edit: Ein Glück müssen wa nun nicht mehr für die zwei Darstellungen (je Tag vs. akkumulierten Gesamtwert) noch einen Satz Queries fahren. :] ]
]