Eine tolle Idee die es gilt umzusetzen!
Peter
Implementierungsplan
Wir sollten uns dem Thema nun kurz und intensiv widmen, da die Bienensaison ja schon bald losgeht und es nicht abzusehen ist, dass wir sonst mittelfristig anderweitig dazu kommen werden.
Unser Vorschlag ist es, ein standalone Programm in Python zu entwickeln, das am besten beschrieben werden kann als: “phenodata is a data acquisition and manipulation toolkit for open access phenology data”.
BBCH Code
Im Rahmen von Forschungsarbeiten zur Grundsteinlegung stoßen wir gerade auf den BBCH Code [1] zur Klassifizierung von Wachstumsphasen, siehe auch [2,3].
USA-NPN
Die Infrastruktur des USA National Phenology Network (USA-NPN) [4] ist ebenfalls nicht von schlechten Eltern. Es bietet offene Daten [5] (auch in Form einer API) sowie ein tolles interaktives Visualisierungstool [6] dazu an.
Weitere Phänologienetzwerke
Da wir die Angelegenheit von der Architektur her betrachtet skalierbar machen wollen, kann uns die Sammlung unter “Phenology Networks Around the World” [7] gut weiterhelfen. Dort sind wir auch auf das “Global Phenological Monitoring” Projekt [8] gestoßen, das im Fachgebiet Agrarklimatologie [9] des Departments für Nutzpflanzen- und Tierwissenschaften an der Lebenswissenschaftlichen Fakultät der Humboldt-Universität zu Berlin angesiedelt ist. Hier wird ebenfalls der BBCH Code [1] zur Klassifizierung verwendet [10].
Feedback welcome!
Relevante Pflanzenarten
P.S.: Eine erste konkrete Frage hätte ich an die Imker unter Euch: Welche konkreten Spezies aus [11] wollen wir denn fürs Erste haben? Also welche Pflanzen sind relevant für die Bienen? Es wäre toll, wenn Ihr dazu eine Liste zusammenstellen könntet, schließlich werden Hafer oder Gerste ja vermutlich nicht dabei sein, stimmts? ;]
[1] BBCH-Code – Wikipedia
[2] http://www.agrometeo.ch/sites/default/files/u10/bbch-skala_deutsch.pdf
[3] http://www.reterurale.it/downloads/BBCH_engl_2001.pdf
[4] https://www.usanpn.org/
[5] Explore Data | USA National Phenology Network
[6] Visualization Tool | USA National Phenology Network
[7] Phenology Networks Around the World | USA National Phenology Network
[8] http://gpm.hu-berlin.de/
[9] Agricultural Climatology — Albrecht Daniel Thaer-Institut für Agrar- und Gartenbauwissenschaften
[10] Phasen
[11] ftp://ftp-cdc.dwd.de/pub/CDC/observations_germany/phenology/annual_reporters/
Weitere Links:
[A] Phänologischer Kalender
[B] Open weather data
[C] https://weather.hiveeyes.org/
[D] Bienenfreundschaft.de - Phänologischer Kalender
Wunschliste relevanter Pflanzenarten
Wir tasten uns gut an die Strukturierung der hier relevanten Details heran. Vielen Dank an @clemens, @peterthiemer, @weef und @wtf für Eure Rückmeldungen zum Thema (folgend Zusammenfassungen aus mails)
Gruppierungen
- primär, “must have”
Die wichtigsten Tracht- und Zeigerpflanzen - sekundär, “nice to have”
Tracht und Zeiger, v.a. lokale oder Imkerpraxis-Bedeutung - tertiär, "unlikely’
Möglicher Erkenntnisgewinn nur sehr schwer und nicht ausschließlich durch pheno-DWD-Daten erlangbar - die können wir hier in den Überlegungen weglassen
Pflanzenarten
Spannend ist der jeweilige Blühbeginn (Pollen- und/oder Nektarlieferant jetzt nicht weiter unterschieden; Unterarten teilweise unterschieden):
-
primär
Angefangen mit dem wichtigsten: die Hasel (jene Pflanze, der wir in unseren Breiten nach der letzten Eiszeit die dauerhafte Existenz der Dunklen Europäischen Biene verdanken). Dann: Schneeglöckchen, Sal-Weide, Löwenzahn, Süßkirsche, Apfel, Winterraps, Robinie,SommerWinter-Linde, Heidekraut. - Damit ist das Bienenjahr abgedeckt (bei ‘uns’). -
sekundär
Dann gibt es noch einige, die der DWD vermerkt und die für Bienen (teils erhebliche) Trachtbedeutung haben und/oder für die imkerlichen Praxis Zeitpunkte markieren. Diese Angaben zu haben, wäre schön - sie haben aber auch schon wieder teilweise nur regionale/lokale Bedeutung:
Wildkirschen (DWD: Kornellkirsche, Traubenkirsche), Stachelbeere, Johannisbeeren, Schlehe, Huflattich, Ahorn, Sonnenblume, Birne, Schwarz-Erle, Roßkastanie -
tertiär bzw. akademisch
Teilweise sehr wichtige, aber schwer zu fassende, nur lokal bedeutsame oder nur durch andere kombinierte Beobachtungen nutzbare Quellen:-
Honigtauquellen
Fichte, Kiefer, Tanne hätte der DWD. Andere infrage kommende Laubbäume ebenfalls (Ahorn, Birke, Birne, Linde, Eiche, Kastanie, Holunder,…).
Die Lebenszyklen der jeweils nötigen Läuse bedingen jedoch, daß aus der Blüte der genannten allein noch keine Tracht ableitbar ist. -
Pheno-Kalender als Hilfe bei möglichen Vergiftungen
Mais, Raps und Sonnenblume. - Wenn gesprüht wird, dann selten im Zusammenhang mit der Blüte, auch ist dann eher der Pollen und seltener der Nektar schlecht, so daß der zeitliche Zusammenhag noch schwieriger zu erkennen wäre.
-
Andere Wünsche
@clemens wünscht sich weiterhin:
- Wir sollten uns auf die “annual_reporters” im Zeitabschnitt “recent” beschränken.
- Ahorn und Roßkastanie würde ich fast schon primär sehen, zumindest in den Städten. Das ist hier die Frühtracht während auf dem Land das Obst dominiert.
- weitere, noch nicht genannte für Imker interessante Pflanzen aus dem DWD-Angebot:
Pflaume, Himbeere, Brombeere, Traubenkirsche, Zweigriffliger Weißdorn - Meine (erweiterte) 10er-Liste ist:
Winterraps, Sonnenblume, Mais, Apfel, Birne, Süßkirsche, Sauerkirsche, Himbeere, Brombeere, Hasel, Heidekraut, Löwenzahn, Robinie, Rosskastanie, Sal-Weide, Spitz-Ahorn, Winter-Linde - Edel-Kastanie, Kornblume, … gibt es nicht, oder?
@peterthiemer wünscht sich noch die Aufnahme des Schwarzen Holunders (u.U. als botanische Zeigerpflanze).
Ich stoße gerade noch auf ein anderes Beobachtungsnetzwerk: naturgucker.de wird vom NABU betrieben. Dieses Jahr findet erstmals eine Kooperation mit dem DWD statt. Im Unterschied zu den sonstigen Beobachtungen des DWD, welche dauerhaft von einzeln bestimmten Ehrenamtlichen durchgeführt werden, wird hier jedermensch aufgefordert, Beobachtungen direkt zu melden.
Mit den genauen Gegenständen der Beobachtung sowie der informationstechnischen Bereitstellung der Daten habe ich mich jedoch nicht befasst – dieser Beitrag soll nur als erster Hinweis auf diese Quelle dienen.
naturgucker mit diy-faktor, das finde ich ja toll: da könnte ich also regelmäßig selbst losstapfen in konzentrischen kreisen um meinen bienenstand, meine beobachtungen bei naturgucker.de hochladen und dann - vorausgesetzt, das lässt sich mit radius um einen ort dort rausziehen - genau meine eigenen beobachtungen (plus das was andere im selben radius um meinen standort eingeben) in den graph importieren. wie gesagt, cool fänd ich das: diese kombination von selber beobachten und in die erfassung und darstellung einfließen lassen.
Du kannst dich auch gerne beim DWD melden. Die suchen noch Beobachter Wetter und Klima - Deutscher Wetterdienst - Der Deutsche Wetterdienst sucht ehrenamtliche phänologische Beobachterinnen und Beobachter
aber der DWD sucht doch nur für bestimmte stellen. in der aktuellen liste mit den gesuchen finde ich leider meine postleitzahl (10999) nicht.
Dürfen wir dazu aufrufen, das “phenodata” Programm in der aktuellsten Version ordentlich durchzutesten und uns Rückmeldungen oder weitere Wünsche zu übermitteln?
Mit dem ersten Schwung wären wir soweit durch, siehe https://github.com/hiveeyes/phenodata/blob/master/TODO.rst.
Danke!
cool. ich mach mich wochenende dran ans testen.
das ist erstmal unabhängig von hiveeyes-grafana, richtig?
ah, ich erinnere mich gerade, die einbindung von wetterdaten läuft ja parallel und ist dann vielleicht recht einfach auch auf die phäno-daten zu übertragen. ich freu mich jedenfalls auf beides.
Danke!
Richtig, wir haben erstmal die Hausaufgaben gemacht, deswegen ist “phenodata” ein komplett standalone Tool geworden. Wir haben natürlich auf der Agenda, die Ereignisse auch ins Grafana einzubinden, aber der Teufel steckt wie immer im Detail und neben der initialen Grafana Lernkurve will das Ganze auch gut durchdacht sein.
Dito! ;]
Zuerst einmal vielen Dank - wieder einmal - für die tolle Arbeit, @Andreas! Win10 hat ja nun auch einen guten Linux-Support und man kann mit “bash” in der Eingabeaufforderung ganz schön Ubuntu nutzen.
Dort habe ich alles recht einfach schon vor ein paar Tagen installieren können, auch das update von phenodata 0.3.0 auf die letzte lief problemlos.
Werden Die DWD-Daten eigentlich immer live vom DWD-Server gezogen oder sind die irgenwo gecachet? Wie gesagft gibt es die DWD-Daten auch schon geografisch als “Grid-Daten” aufbereitet. Da müssen wir einmal schauen, ob wir und die nächste Station dann selbst suchen oder ob es Vorteile hat die Grid-Daten zu nutzen, da die z.B. schon berücksichtigen, dass zwischen meinem Standort und der nächsten Station ein Höhenzug liegt, der Unterschied könnte aber auch Erbsenzählerei sein. Ich werde mir auf jeden Fall noch die verschiedenen Datenquellen genauer ansehen, die aktuellen vs. “historic” und die Jahresmelder vs. Sofortmelder.
Die Vorhersage müssen wir uns genau anschauen, ich finde es kritisch, wenn wir Daten die aktuell reinkommen als “Prognose” verwenden. Wenn das direkt neben dem eigenen Stand ist mag das klappen, wenn das in Franke ist und ich auf der Schwäbschen Alb, dann nicht. Der Mittelwert der letzten Jahre vom gleichen Standort geht sicher. Wenn die Meldungen aber erst reinkommen muss man checken ob die schon halbwegs vollständig sind. Der DWE gibt dann immer an von wie viel Prozent der Melder Daten schon vorliegen.
Wir (@Andreas und ich) haben uns die Tage einmal bilateral verständigt und überlegt, welche Daten des DWD für einen Imker als Daten für “seine” Standort herangezogen werden können. Es könnte
- die räumlich nächstgelegene Meßstation herangezogen werden oder
- die “Grid-Daten” des DWD.
Die Grid-Daten (Beschreibung: ftp://ftp-cdc.dwd.de/pub/CDC/grids_germany/annual/phenology/BESCHREIBUNG_gridsgermany_annual_phenology_de.pdf), FTP-Zugang zu den Daten: ftp://ftp-cdc.dwd.de/pub/CDC/grids_germany/annual/phenology/) sind geografisch aufbereitete Daten (Auflösung 1 km x 1 km), bei denen die (vermuteten) Werte für einen Bienenstand aus den Werten der umgebenden Messstationen berechnet wird. Das Verfahren beschreibt der DWD so, häutsächlich eine lineare Regresseion bei denen die Parameter geographische Lage (Länge, Breite) und Höhe über NN berücksichtig werden:
Im Dokument steht auch, welche Pflanzen dabei berücksichtigt werden, es kann sein, dass es nicht alle sind, die erhoben werden; weiter werden Qualitätsparameter nicht berücksichtigt - was immer das bedeuten möge. Todo für mich wäre
- Eine Karte der bestehenden Messstationen erstellen, um zu schauen, wie diese regional verteilt sind. Wenn diese sehr weit auseinander sind 100, 200 km macht die nächste Station als Datenquelle für den eigenen Stand wenig(er) Sinn und die Grid-Daten wären bevorzugt. – Falls die Stationen engmaschig und gleich verteilt sind, könnte man auch auf die nächste Station zugreifen.
- Abgleich, ob in dern Grid-Daten z.B. Pflanzen fehlen oder wir die Qualitätsmarker brauchen, die nicht in den Grid-Daten vorhanden sind.
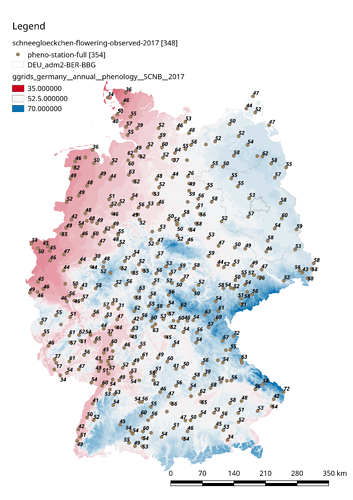
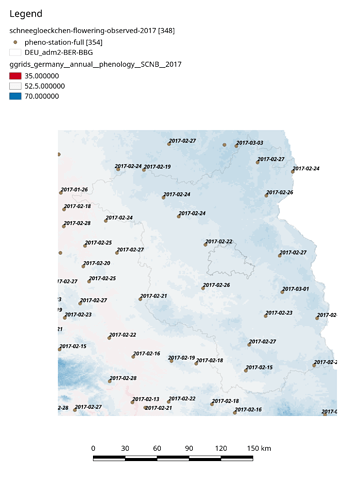
354 davon gab es 2017, Schneeglöckchen-Blüte haben davon 348 gemeldet. In der oberen Karte ist das Ereignisdatum abgetragen, in der unteren der “Jultag”, also das Ereignis als Tag des Jahres (die Legenden der Karten sind überarbeitungswürdig, ich weiß). Der zweifarbige Gradient sind keine Höhenzüge, sondern ist der “Jultag” als gemapter Rasterwert:
Gerade Berlin/Brandenburg hat nicht allzuviele Jahres Sofortmelder, überall woanders gibt es mehr:
Tolle Bilder, wow!
Wurden die Karten aus den o.g. Grid-Daten generiert? Wir kamen bisher noch nicht dazu, darauf einen Blick zu werfen, sollten wir sie ebenfalls per “phenodata” erschließen?
ja, die sind aus den grid-Daten gebaut; das textuelle overlay ist mittels Deiner phenodata 0.6.4 erzeugt.
Die Grid-Daten [1] sehen ja vogelwild aus, danke ;]. Wenn Du uns auf die Sprünge helfen kannst, wie man die am schlauesten interpretieren kann, sehen wir, was wir tun können. Können wir die Erzeugung der Bilder insgesamt irgendwie automatisieren?
[1] ftp://ftp-cdc.dwd.de/pub/CDC/grids_germany/annual/phenology/SCNB/grids_germany_annual_phenology_SCNB_2017.asc.gz
Auf der Karte schaut es ja so aus, als ob Berlin gar keine Station hat. Welche query hast du denn benutzt? Habe es mal mit
phenodata nearest-stations \
--source=dwd --dataset=annual \
--latitude=52.518611 --longitude=13.40833
versucht. Geo-Koordinaten sind die von Wikipedia für Berlin, ungefähr Rotes Rathaus und da bekommt man u.a.
12132 Berlin-Dahlem
12130 Berlin-Marienfelde
12135 Berlin-Frohnau
13961 Berlin-Zehlendorf
15556 Berlin-Köpenick
15208 Berlin-Rahnsdorf
das: phenodata list-stations --source=dwd --dataset=annual --station=berlin funktioniert übrigens nicht (--station=berlin wird nicht honoriert). Und phenodata list-stations --source=dwd --dataset=annual --sort=Stationsname auch nicht (hier ist das Problem --sort=Stationsname)
diesen hier - ermittelt mir alle, die in 2017 für Objekt 127 (Schneeglöckchen) das Ereignis 5 (Blühbeginn) gemeldet haben:
$ phenodata observations --source=dwd --dataset=immediate --filename=Schneegloeckchen --partition=recent --year=2017
2018-03-21 22:10:37,954 [phenodata.dwd.pheno ] INFO : Scanning for files
2018-03-21 22:10:37,955 [phenodata.ftp ] INFO : The cache directory is /home/msw/.cache/phenodata/dwd-ftp
2018-03-21 22:10:37,960 [phenodata.dwd.pheno ] INFO : Starting data acquisition with 1 files
100%|█████████████████████████████████████████████| 1/1 [00:00<00:00, 47.94it/s]
2018-03-21 22:10:37,982 [phenodata.dwd.pheno ] INFO : Entering flux compensator: Filter and transform data
+---------------+----------------+--------------------+-------------+------------+------------------+---------------------+----------+
| Stations_id | Referenzjahr | Qualitaetsniveau | Objekt_id | Phase_id | Eintrittsdatum | Eintrittsdatum_QB | Jultag |
|---------------+----------------+--------------------+-------------+------------+------------------+---------------------+----------|
| 140 | 2017 | 1 | 127 | 5 | 2017-02-25 | 1 | 56 |
| 164 | 2017 | 1 | 127 | 5 | 2017-02-26 | 1 | 57 |
| 183 | 2017 | 1 | 127 | 5 | 2017-02-16 | 1 | 47 |
| 198 | 2017 | 1 | 127 | 5 | 2017-02-27 | 1 | 58 |
| 596 | 2017 | 1 | 127 | 5 | 2017-02-17 | 1 | 48 |
| 662 | 2017 | 1 | 127 | 5 | 2017-02-19 | 1 | 50 |
| 853 | 2017 | 1 | 127 | 5 | 2017-02-27 | 1 | 58 |
| 1412 | 2017 | 1 | 127 | 5 | 2017-02-23 | 1 | 54 |
[...]Also
$ phenodata observations --source=dwd --dataset=immediate --filename=Schneegloeckchen --partition=recent --year=2017 --format=csv | wc -l -
[...]
349-minus eine csv-header-Zeile macht 348 Meldungen , wie in den Grafiken oben auch.
Die Gesamtzahl der Stationen oben stammt aus (das entstandene file ist dann im GIS gelandet):
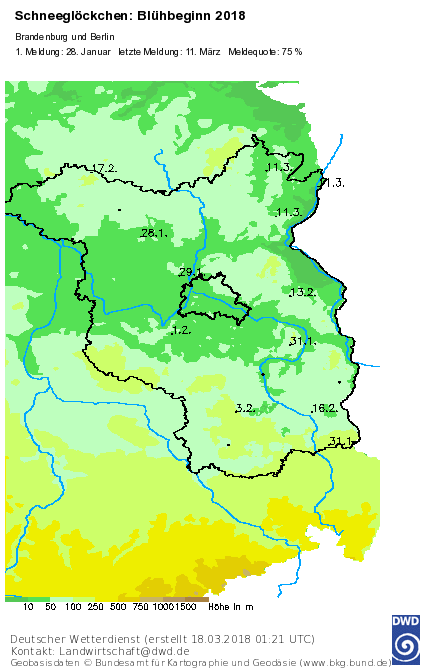
phenodata list-stations --source=dwd --dataset=immediate --format=csv >pheno-station-full.csvFür dieses Jahr sieht das beim DWD so aus, die bislang letzte Meldung stammt vom 11.März 2018
(Achtung: Farbgradient bedeutet hier Höhenzüge, kein gemapter Blühbeginn wie oben!) - was die Anzahl und Position der Melder angeht, kann ich insofern Deine Beobachtung, @clemens, nicht bestätigen:
na, mit ner GIS; in diesem Fall QGIS 2.18.17 .